獻給所有斜槓生物資訊的人們
程式設計大概是世界上最容易在網路上找到答案的領域了,奇妙的是有關於生物資訊(生資)的程式設計與分析,在中文的世界卻寥寥無幾。對從 Wet Lab (生物實驗) 跳到 Dry Lab (生資分析) 的你來說,學習充滿邏輯的程式語言是一個坑,學習充滿生物意義的資料結構更是一個坑。
而從資訊領域跳到生資分析的你來說,看著莫名其妙跟我不熟的序列與生物知識,難懂 Wet Lab 的他們想要我把資料視覺化成什麼樣子。
而下列的式子仍舊是不變的道理 :
程式=演算法+資料結構
只是不小心撒上了一些生物 : 生資分析=生物意義+演算法+資料結構
於是本系列文章就這麼誕生了! 以 16S rRNA 微生物組定序分析為基礎,追完這些文章或許沒辦法讓你變成高手,但是希望能讓你獲得最最最基礎的能力,在充滿原文的知識海裡遨遊時能更得心應手。
在開始前我需要哪些先備知識
本系列以指令操作分析為主,先學習使用別人的程式,待你熟悉分析的產物之後,就能站在巨人的肩膀上,再自行學習 Python, R, MATLAB 等技能,讓資料的視覺化更完美。
1. 基礎Linux基本指令
通常生資分析所需要的運算資源甚多,Linux純指令介面少了圖形化介面的資源運算,運算效率較佳,適合大量運算的工作,實務上多半以遠端連線到 Linux 伺服器/工作站。畢竟你家通常不會有一台好幾十萬的電腦,我說通常。
- 我知道 Linux 跟 Windows 都是作業系統
- 我知道 如何連上遠端 Server
- 我知道 如何將檔案上傳到遠端 Server、下載資料到本機端
- 我知道 什麼是 cd, mv, cp, mkdir, rm, pwd, vim
- 我知道 什麼是 絕對路徑與相對路徑
2. 生物知識
至少需要有高中選修生物或大一普通生物學的基礎知識。
- 我知道 DNA 是雙股螺旋、DNA電泳、PCR, Real-Time PCR 原理
- 我知道 核醣體 (Ribosome)、mRNA, tRNA, rRNA
- 我知道 轉錄 (Transcription)、轉譯 (Translation)
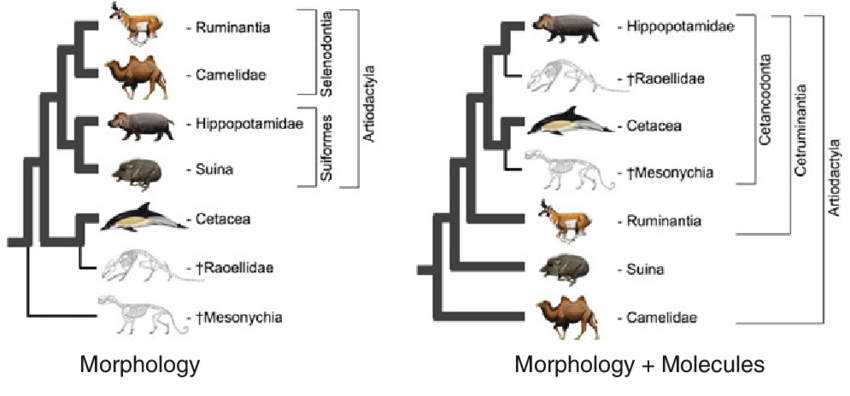
- 我知道 親緣關係樹 (Phylogenetic Tree) 可以依據分生(Molecules)與型態(Morphology)繪製
3. Git (git clone) 與 GitHub
說要會 Git 版本控制 (Version control) 就太殘忍了,至少要會以下事情 :
- 我知道 GitHub 有地球上所有工程師心血結晶 – 滿滿的程式資源
- 我知道 git clone 指令,會下載 GitHub 上的 Package
4. Google
- 做實驗失敗可能在這找不到答案,但做生資請一定要會用它
學習地圖
學習地圖的存在可以幫助你更了解整個知識架構 :
- [Day 21] 統整 NGS 次世代定序 16S rRNA 生資分析學習地圖
- [Day 28] 統整 TGS 第三代定序 16S rRNA 生資分析學習地圖
看完系列文我會些什麼 ?
- 知道定序技術是什麼
- 知道如何分析 16S rRNA 次世代定序 (Next Generation Sequencing, NGS)
- 知道如何分析 16S rRNA 第三代定序 (Third Generation Sequencing, TGS)
- 知道 16S rRNA 定序需要的資料視覺化有哪些方式
- 知道程式設計在生資的角色
- 可能會知道自己適不適合成為生資人