定序完後的資料在哪裡 ? – SRA (Sequence Read Archive)
前一回我們探討了 NGS 從 DNA 樣本到上機 (上 Illumina 機器定序),今天找找前人上機後的資料,究竟都去哪裡了~ 如果已經有一批定序資料下載好等著你練習了,那麼就往下看下一篇XD
SRA (Sequence Read Archive) 資料庫存放全世界研究的定序資料,基本上要發有關定序資料分析結果的 Paper 通常都會要求上傳到這兒,包含 RNA seq、Single cell sequencing、Whole genome sequencing、Metagenomic、16S rRNA 等,通常–>就代表很多人不乖。
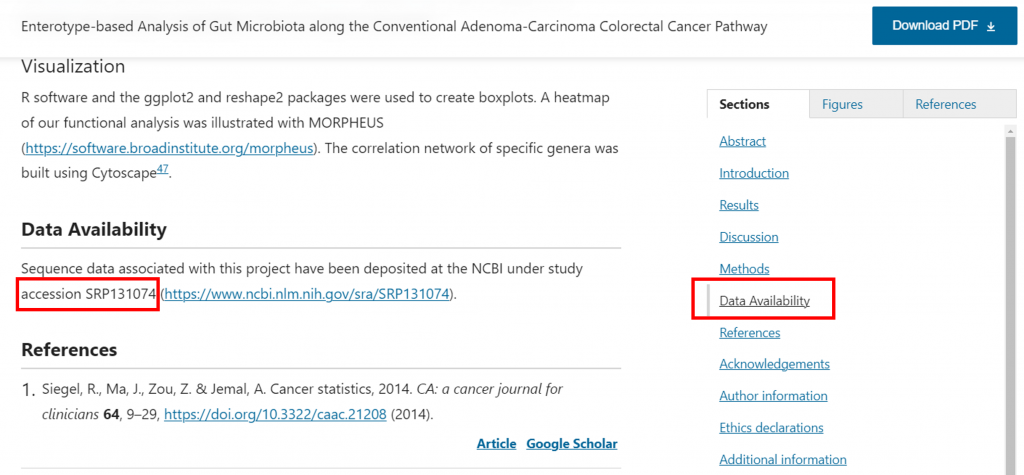
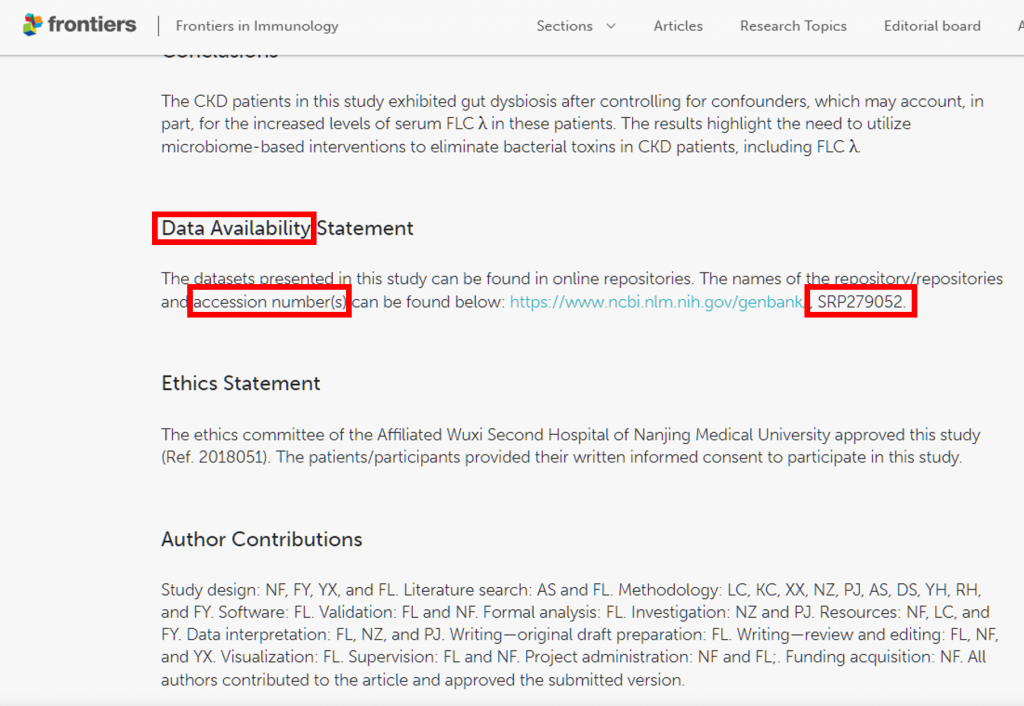
從文獻取得 accession number 首先,得先在 Paper 中拿到一串 accession number,有上傳到 SRA 的 Paper 則會在文中以: Data Availability、repositories、accession number、accession SRPXXXXXX 關鍵字出現:
帶著 Accession number 去餵 SRA Run Selector
以範例 1 來說,帶著 SRP131074 餵給 SRA Run Selector:
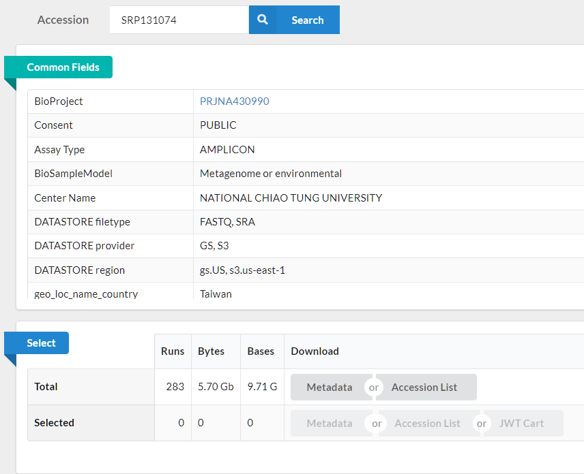
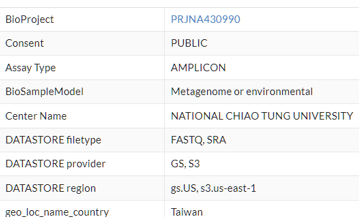
在 Common Fields 中確認基本資訊是否與文獻提及的內容相符 (檢體類型、序列類型等),通常我還會確認是否有 Platform: ILLUMINA、LibraryLayout: PAIRED、metagenome 等關鍵字。
挑選想分析的序列資料
往下滑會看到表格,從 Library Name、Sample Name推敲每個序列檔案代表的資訊,通常會與 Paper 中分組的代號相關,好心一點會放在文獻的 Supplementary Material,實務上很多都寫得不清不楚,所以需要一點推敲,在這裡我勾擇 CC,代表 已經發展成大腸癌的患者檢體 (Colorectal Cancer):
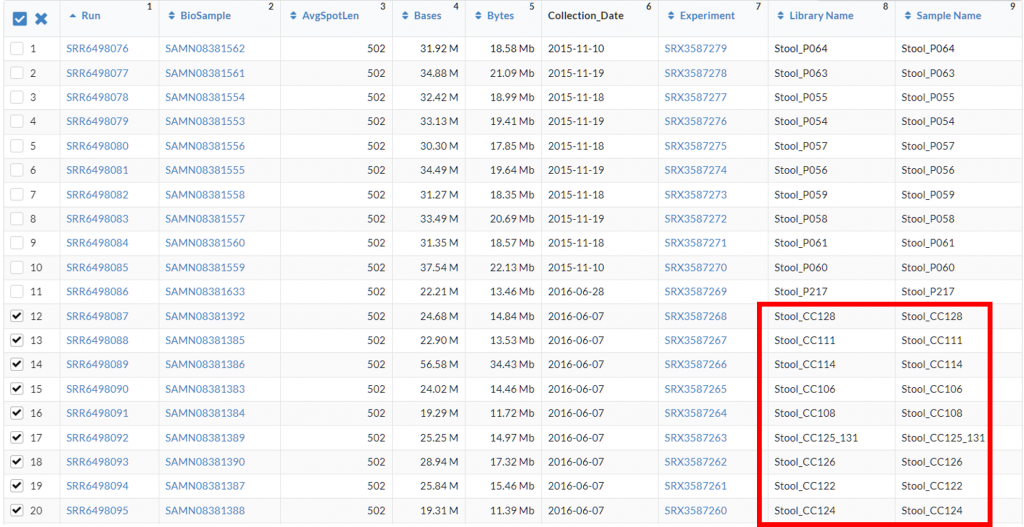
下載 Accession List 勾選後,可以往上滑點 Accession List 下載每個序列檔案的代號:
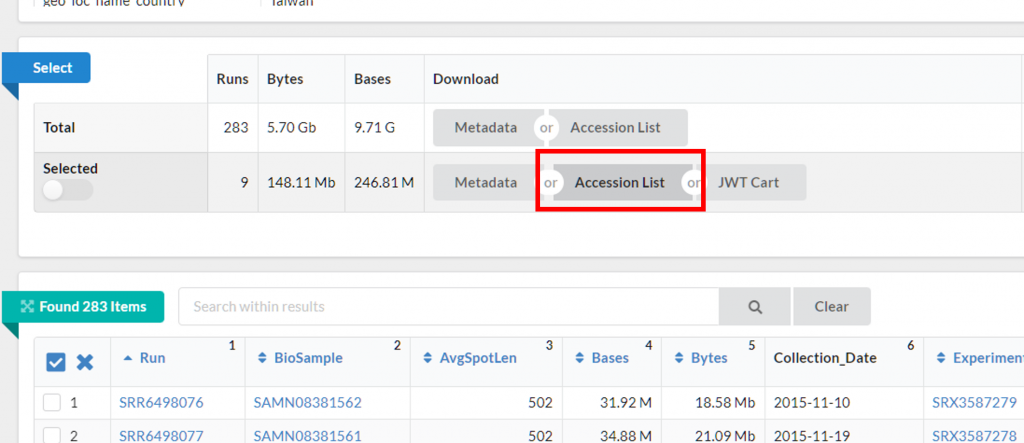
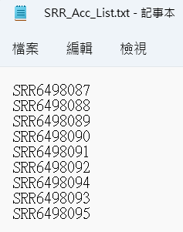
後續 SRA Toolkit 會根據 Accession List 檔案抓取資料,偷偷點開下載的檔案,其實很單純,就是剛剛勾選的序列檔案代號。
安裝序列下載工具 Python bioinfokit & SRA Toolkit
現在帶著 Accession List 要去 Linux 準備下載序列資料啦! 16S rRNA 序列檔案大小雖然較 RNA seq 小,但是樣本數一多還是很可觀的,SRA Toolkit 提供使用者可以快速下載 NCBI SRA 資料庫上的序列資料,會依照你給的 Accession List 到 SRA database 下載資料。但是對於一次需要下載數十個甚至數百個定序資料分析,就需要 Python bioinfokit 提供批量下載的功能。
16S 的資料分析往往需要好幾十個以上的樣本菌相比較,才能看出整體菌相差異性。
下列流程是預設使用者使用 Linux,並已安裝完 Anaconda 或 Miniconda
批量下載定序資料 – Python bioinfokit
先用 conda 創造一個虛擬環境,避免跟原本的你使用的 python 環境打架。
- 可以把 conda 想成一種包含 Python 與各類幫助分析套件的軟體,在 conda 建立虛擬環境 (就像是沙盒),可以幫助我們從事不同的分析使軟體彼此不衝突,例如: 下載序列軟體放在一個沙盒、分析序列軟體放在另一個沙盒。
conda create --name SRA_download python==3.8會顯示的畫面 (以 miniconda 為例) :
[XXXXXX@localhost]$ conda create --name SRA_download
Collecting package metadata (current_repodata.json): done
Solving environment: done
## Package Plan ##
environment location: /home/XXXXXX/miniconda3/envs/SRA_download
Proceed ([y]/n)?輸入 y 後 Enter,會顯示的畫面 :
Preparing transaction: done
Verifying transaction: done
Executing transaction: done
#
# To activate this environment, use
#
# $ conda activate SRA_download
#
# To deactivate an active environment, use
#
# $ conda deactivate創造完之後,輸入以下指令進入 SRA_download 虛擬環境 :
conda activate SRA_download成功的話會在你的名稱前面出現 (SRA_download) :
(SRA_download) [XXXXXX@localhost ~]$安裝 bioinfokit python 套件:
conda install -c bioconda bioinfokit輸入 y 後 Enter,會經歷一段漫長的過程(?,直到看到這三行出現就對了~
Preparing transaction: done
Verifying transaction: done
Executing transaction: done再裝這些套件:
pip install NumPy scikit-learn seaborn pandas matplotlib SciPy matplotlib_venn adjustText statsmodels tabulate textwrap3跑出 Successfully 的相關訊息就全部安裝完成拉~
'Successfully' built matplotlib_venn adjustText
Installing collected packages: textwrap3, pytz, threadpoolctl, tabulate, six, pyparsing, pillow, NumPy, kiwisolver, joblib, fonttools, cycler, SciPy, python-dateutil, patsy, packaging, scikit-learn, pandas, matplotlib, statsmodels, seaborn, matplotlib_venn, adjustText
'Successfully' installed NumPy-1.23.2 SciPy-1.9.1 adjustText-0.7.3 cycler-0.11.0 fonttools-4.37.1 joblib-1.1.0 kiwisolver-1.4.4 matplotlib-3.5.3 matplotlib_venn-0.11.7 packaging-21.3 pandas-1.4.4 patsy-0.5.2 pillow-9.2.0 pyparsing-3.0.9 python-dateutil-2.8.2 pytz 2022.2.1 scikit-learn-1.1.2 seaborn-0.11.2 six-1.16.0 statsmodels-0.13.2 tabulate-0.8.10 textwrap3-0.9.2 threadpoolctl-3.1.0定序資料工具 – SRA Toolkit
下載 3.0.0 版本 SRA Toolkit 壓縮安裝檔到本機:
wget https://ftp-trace.ncbi.nlm.nih.gov/sra/sdk/3.0.0/sratoolkit.3.0.0-ubuntu64.tar.gz解壓縮安裝檔:
tar -zxvf sratoolkit.3.0.0-ubuntu64.tar.gz你在的目錄會多一個 sratoolkit.3.0.0-ubuntu64 資料夾,就是主程式。接著設定環境變數:
export PATH=$PATH:$PWD/sratoolkit.3.0.0-ubuntu64/bin調整預設設定
vdb-config -i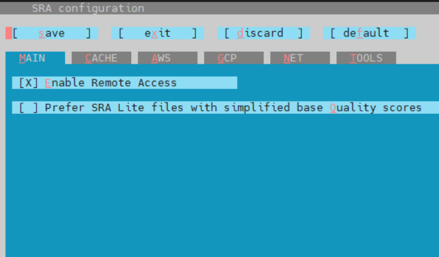
紅色底線的字母就代表進入需要按的按鍵:
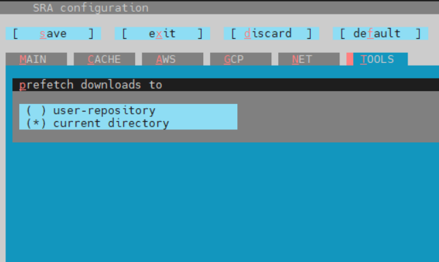
按 T 進入 TOOLS,再按 P ,上下鍵選擇 current directory,再按 S 儲存,再按 o 代表 ok ,按兩次 Esc 即可離開。
下載小資料試試
你的目錄應該要有:
- 最一開始下載的檔案
SRR_Acc_List.txt,將他放到 Linux 目錄,
這邊也提供範例檔案包含六種序列SRR_Acc_List.txt:
SRR6498087
SRR6498088
SRR6498089
SRR6498090
SRR6498091
SRR6498092- 接著我們製作一個 python 執行檔,檔名可自訂,教學方便叫做
batch_download.py,內容如下 (可以在 Linux 以 vim 創建或是在 Windows / Mac 建好傳上去)
from bioinfokit.analys import fastq
fastq.sra_bd(file='SRR_Acc_List.txt', t=16, other_opts='--split-files' )t=16,代表使用 16 核心file='SRR_Acc_List.txt',代表包含序列編號的檔案other_opts='--split-files',代表輸出的檔案要將Forward Reverse分別以_1, _2 表示
執行 batch_download.py:
python batch_download.py會顯示的畫面:
Downloading SRR6498087
Downloading SRR6498088
Downloading SRR6498089
Downloading SRR6498090
Downloading SRR6498091
Downloading SRR6498092 等到下一行再次出現,就代表完成啦 !
[XXXXXX@localhost ~]$可以 ll 查看目錄是否有該檔案:
-rw-r--r-- 1 XXXXXX users 28171694 Aug 23 15:10 SRR6498087_1.fastq
-rw-r--r-- 1 XXXXXX users 28171694 Aug 23 15:10 SRR6498087_2.fastq
-rw-r--r-- 1 XXXXXX users 26139160 Aug 23 15:11 SRR6498088_1.fastq
-rw-r--r-- 1 XXXXXX users 26139160 Aug 23 15:11 SRR6498088_2.fastq
-rw-r--r-- 1 XXXXXX users 64697336 Aug 23 15:13 SRR6498089_1.fastq
-rw-r--r-- 1 XXXXXX users 64697336 Aug 23 15:13 SRR6498089_2.fastq
-rw-r--r-- 1 XXXXXX users 28171694 Aug 23 15:10 SRR6498090_1.fastq
-rw-r--r-- 1 XXXXXX users 28171694 Aug 23 15:10 SRR6498090_2.fastq
-rw-r--r-- 1 XXXXXX users 26139160 Aug 23 15:11 SRR6498091_1.fastq
-rw-r--r-- 1 XXXXXX users 26139160 Aug 23 15:11 SRR6498091_2.fastq
-rw-r--r-- 1 XXXXXX users 64697336 Aug 23 15:13 SRR6498092_1.fastq
-rw-r--r-- 1 XXXXXX users 64697336 Aug 23 15:13 SRR6498092_2.fastq 如果發現檔案名稱並非 .gz 副檔名結尾的壓縮檔,記得將檔案做壓縮,避免後續分析報錯。
gzip *.fastq到這裡都順利,可以將 sratoolkit.3.0.0-ubuntu64.tar.gz 殘餘的安裝檔刪除。
rm sratoolkit.3.0.0-ubuntu64.tar.gz下次需要下載序列資料時
只要啟動虛擬環境就可以從本篇的「下載小資料試試」,就能繼續分析新的資料。
conda activate SRA_download而離開環境的方式:
conda deactivate就會看到前面的括號環境名稱消失,就是成功離開。