尋找各組別的 Biomarker 菌種
為方便後續講解我們做個小約定,不同階層的分類名稱之為分類單元,例如菌物界為一分類單元、擬桿菌門為一分類單元。
儘管 Alpha 與 Beta 多樣性分析可以一覽組內與組間的特徵,若想要一窺究竟是哪些分類單元在組別間數一數二發光發熱,LEfSe 就是一款以統計分析為基礎,在組別間進行比較,獲得各組別微生物生物標記 (Biomarker) 的軟體。
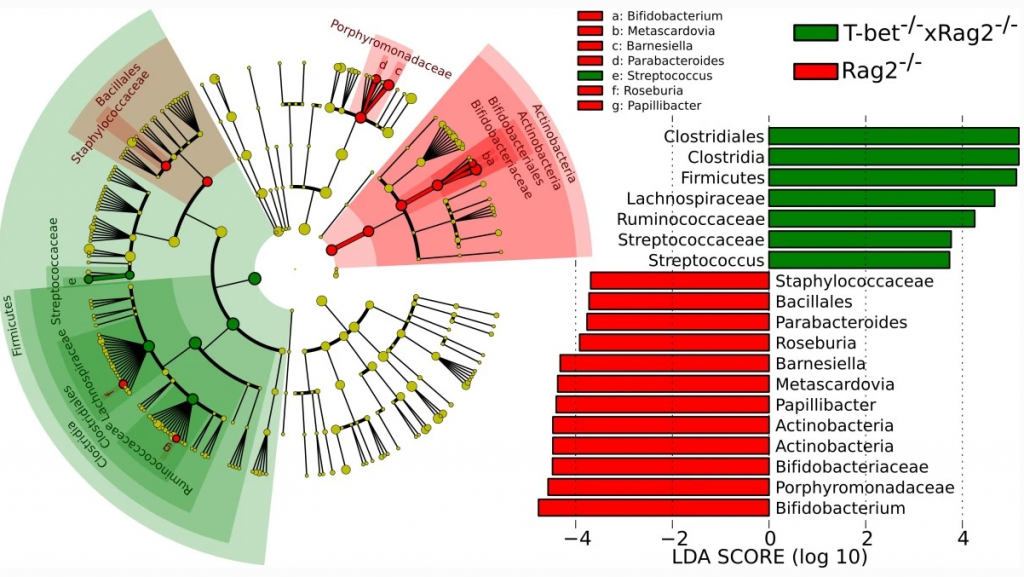
(Segata, Nicola, et al., 2011)
LEfSe 分析原理三部曲
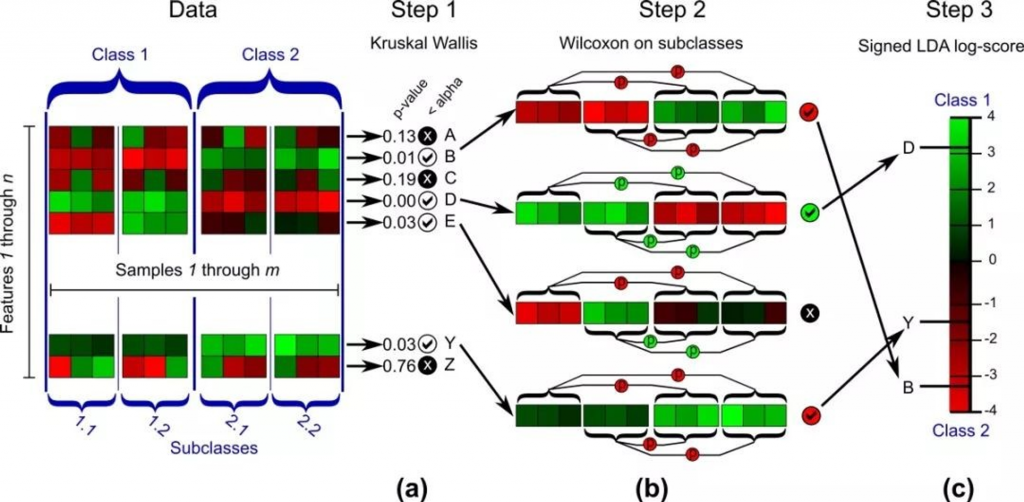
1. 挑選出多組間豐富度有差異的菌種們 (初賽)
Kruskal-Wallis 多樣本中位數差異檢定。
在不同組別的之中,想知道多組的樣本間該菌種是否有差異,LEfSe 第一步使用了Kruskal-Wallis檢定,此檢定為無母數方法,樣本數較小 (小於30),且適合欲分析對象含有三組以上樣本,最終得出具有顯著差異之候選菌種們,在上圖 (a) 的例子中B, D, E, Y 通過了初賽。
2. 將有差異的菌種們於兩組間相互比較 (複賽)
Wilcoxon 雙樣本中位數差異檢定。
得出具有顯著差異之候選菌種們之後,進入兩兩組別互相廝殺的賽制中,LEfSe 第二步使用了 Wilcoxon 檢定,該檢定同樣為無母數方法,樣本數較小 (小於30),且適合欲分析對象為兩組樣本相互成對,最終得出兩兩比較後仍有顯著差異晉級之候選菌種們,在上圖 (b) 的例子中B, D, Y通過了複賽。
3. 評估候選菌種的影響力 (決賽並排名)
LDA (Linear Discriminant Analysis) 線性判別分析。
晉級決賽的候選菌種們,最後要進行評估此菌種在組中的影響力,並化為分數進行排名 (LDA score)。LDA 與 先前提及 Beta Diversity 中 PCA 相似,都是透過降維方式了解各樣本間的相似程度,但 PCA 使用的是非監督式學習 (unsupervised learning)。而 LDA 採用的是監督式學習 (supervised learning),想不到這裡也藏了機器學習 (?) 可參考 Tommy 大大文章。
用個超級輕鬆講法就是 LDA 相對於 PCA ,在請電腦做降維前,先告訴它 :「嘿嘿我們哪些樣本是屬於哪一 組(告知分組狀態)」,電腦之後就能比較清楚的劃分組間的界線,留下更容易區分各組的維度,在上圖 (c) 的例子中將B, D, Y 量化顯著差異的程度。
所需的輸入檔案
- 分析前的註釋檔案 sample-metadata.tsv [第 06 篇]
- 品質管制後的檔案 table-dada2.qza [第 08 篇]
- 物種分配後的檔案 taxonomy.qza [第 11 篇]
所需的環境
LEfSe 所吃的輸入檔案比 [第 19 篇] PICRUSt2 所需的檔案格式複雜且傲嬌,有位偉大的前輩受不了製作輸入檔案的痛苦,開發了 dokdo 套件拯救芸芸眾生。在啟動 QIIME2 環境的前提下,安裝 dokdo 套件,幫助製作 LEfSe 所需的輸入檔案。
測試發現,QIIME2 版本需在 2022.2 (含)以下才能順利安裝 dokdo,因此本篇文章以QIIME2 2022.2 版本教學。
安裝 QIIME2 2022.2
wget https://data.qiime2.org/distro/core/qiime2-2022.2-py38-linux-conda.yml
conda env create -n qiime2-2022.2 --file qiime2-2022.2-py38-linux-conda.yml啟動 QIIME2 環境
conda activate qiime2-2022.2安裝 dokdo 套件
conda install -c hcc dokdo下回會介紹分析的實作 !