透過 dokdo 製作 LEfSe 輸入資料
在已經裝好 dokdo 的 QIIME2 環境下輸入,不需要跑很久,通常一兩分鐘內會完成 :
dokdo prepare-lefse \
-t table-dada2.qza \
-x taxonomy.qza \
-m sample-metadata.tsv \
-o input_table.tsv \
-c Sex
#若不在目前所在資料夾,記得加入相對路徑or絕對路徑告知檔案位置。dokdo prepare-lefse: 使用 dokdo 套件中 prepare-lefse 函式
-t / -x / -m 分別為需要的資料。
-o 則是產出的檔案名稱。
-c 是填入想要分析的組別,務必與 sample-metadata 所描述的組別名稱完全相同。
本篇教學為最基礎的 LEfse 操作,省略了 subclass subject 複賽參數的設定,如果想要加入可以參考官方文件。
可能出現的報錯情況 :
raise KeyError(key) from err
KeyError: 'group'代表組別名稱輸入錯誤,可以藉由查看 sample-metadata 確認組別名稱:
vim sample-meatadata.tsv完成後不會顯示任何成功訊息,可以查看輸出的資料夾有沒有對應的檔案名稱,input-table.tsv。
Galaxy with LEfSe 網頁版
秉持能少一事就少一事的精神 (?),這邊使用 Galaxy 網頁內含的 LEfSe 線上分析功能 :
1. 點上傳按鈕 :
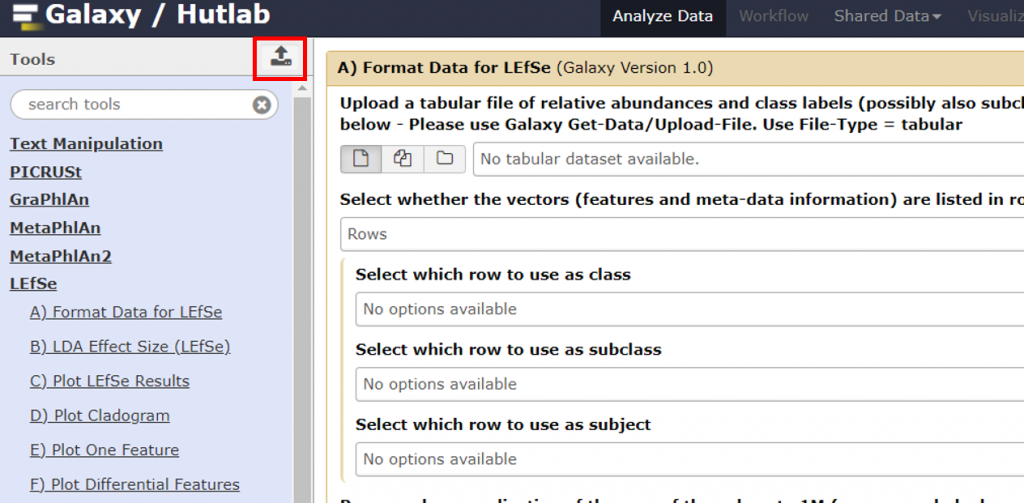
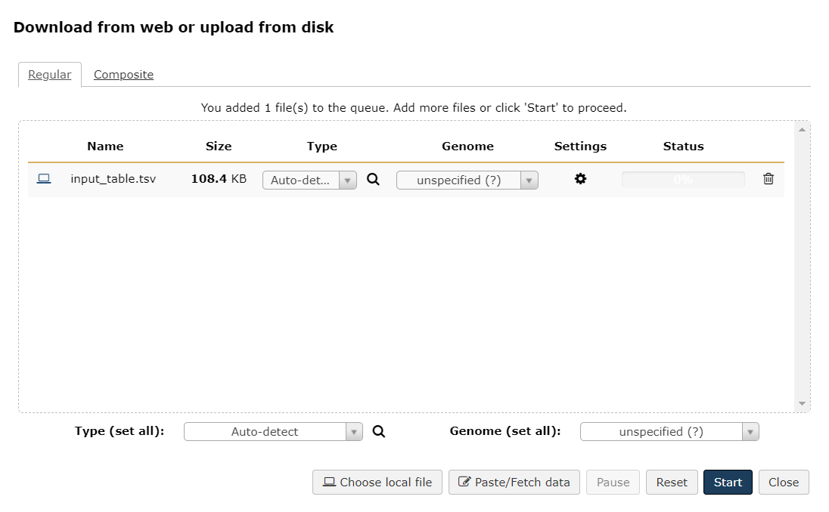
2. 上傳 input-table.tsv 檔案,記得在右下角按 Start
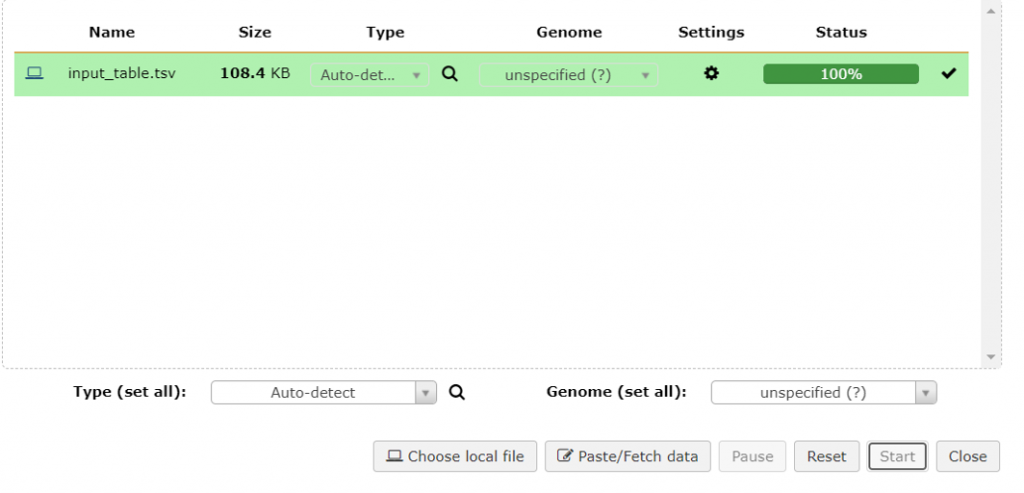
3. 看到舒服的綠燈就可以按右下角 Close
4. 點左側 LEfSe > A) Format Data for LEfSe
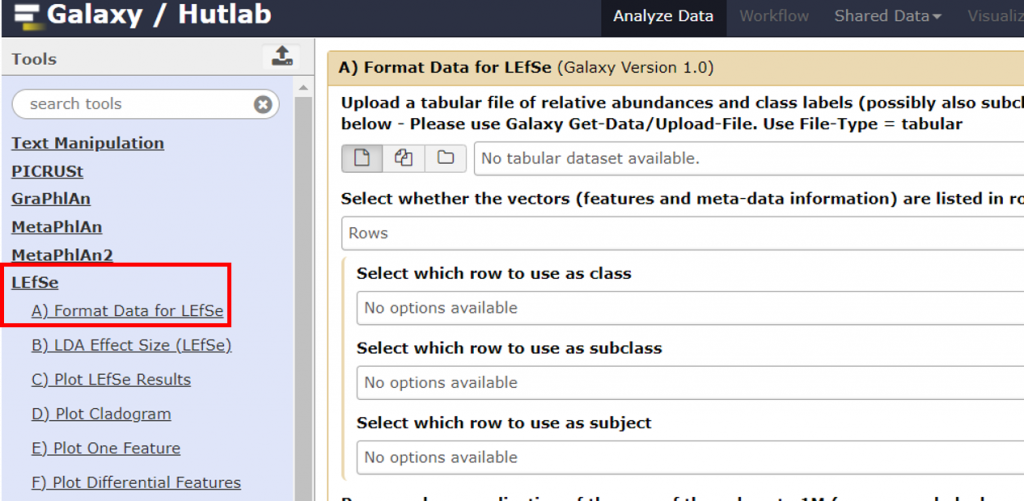
5. 會顯示剛剛的檔案,若紅框處沒有出現剛剛上傳的檔案名稱,可以再點擊 LEfSe > A) Format Data for LEfSe 或重整頁面。在這邊會有一些參數的調整,教學方便都以預設值不變動,之後長大 (萬一不小心簽博了) 想做點進階分析就可以調整 SubClass 與 Subject。
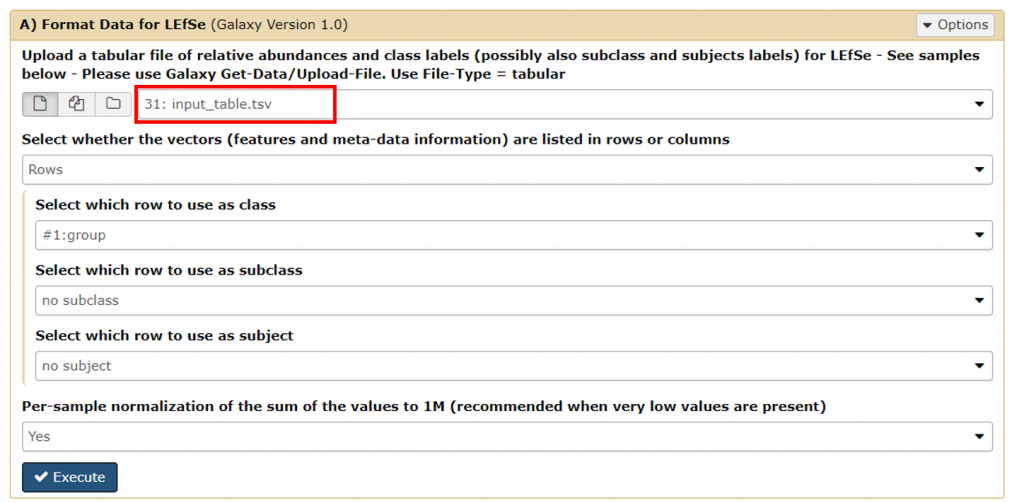
6. 無懸念地點下 Execute,出現綠色視窗請給自己一個掌聲! 並點 B) LDA Effect Size (LEfSe) 進行排名分析。
7. 這邊可以設定包含各步驟統計分析的 p value (熟悉的 0.05) 以及 LDA score Log 值 (default 2),如果發現預設跑不出顯差Q,可以考慮在 Set the strategy for muti-class analysis 選項中選擇 One-against-all(less strict) : )

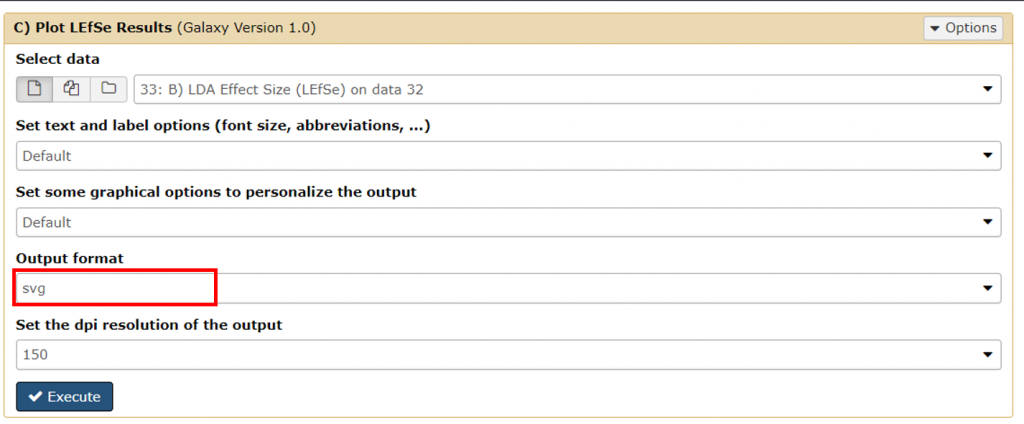
8. 同樣會出現綠色視窗,請繼續為自己歡呼,點 C) Plot LEfSe Results,要產圖囉! 在此是 LDA 的橫向柱狀圖,這邊有多種解析度 dpi /字型等參數可以調整,因為下一篇有調整顏色的教學,因此這邊格式選擇 svg。
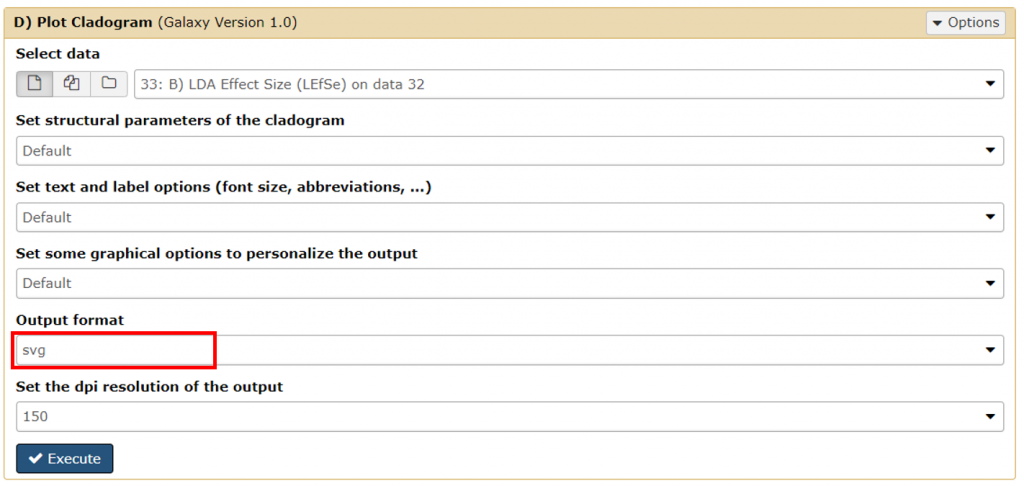
9. D) Plot Cladogram 則是可以產出的經典 LEfSe 同心圓,同樣可以做基本的產圖調整,一樣先選擇 svg。
10. 注意到右側有曾經上傳的檔案以及產出的檔案,可以在想要下載的檔案點檔案名稱就會跑出可以儲存的圖案
11. 點開後就可以用瀏覽器查看圖片了~ (這邊先放 LDA 示意,同心圓就交給讀者點開了)
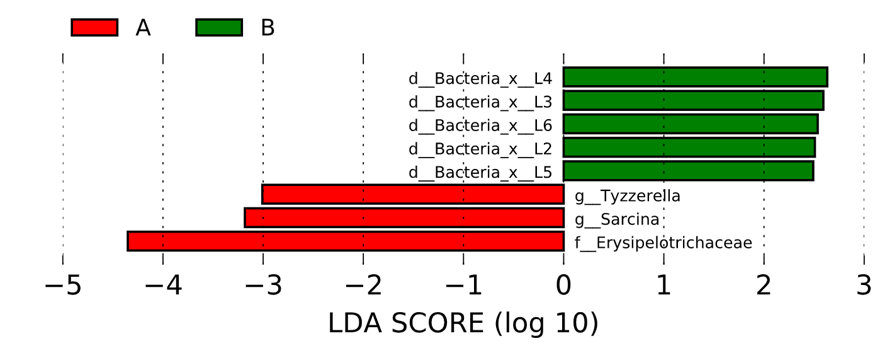
到此就獲得 LEfSe 的結果,如果發現錯誤訊息關於無顯著差異也不要難過,分析沒有結果也是一種結果,而若有幸運產出圖則可以開始試著查看顯著差異的菌種資訊與文獻,說不定有意想不到與你研究有關連結。
其實 LEfSe 有提供 CLI 指令式操作,但筆者本機疑似相容性與版本衝突問題,暫時沒有解決,改以網頁版工具呈現,效果相同。
下回會介紹顏色的調配! 典型的 LEfSe 即是你所看到的上述模樣,身為點了一些前端網頁工程技能的筆者,看到這該死的配色實在是有點美感焦慮,於是我們來動動手腳,讓研究結果的呈現更有美感~